La Revista Memorias del Instituto Osvaldo Cruz (FIOCRUZ) de Brasil publicó un nuevo artículo del Grupo de trabajo sobre SARS-2 y Covid-19 formado por investigadores de las Secciones Genética Evolutiva y Virología de la Facultad de Ciencias y el Departamento de Laboratorios de Salud Pública (DLSP - MSP)
 En su camino evolutivo de adaptación al hospedero humano, el SARS-CoV-2 genera diversos cambios genómicos. Entre ellos encontramos las mutaciones puntuales, que cambian una sola de las bases del genoma, y las inserciones y deleciones (llamadas también “indels”), que agregan o quitan bases. Estos cambios ocurren en todo el genoma, incluso en los genes accesorios u ORFs, los cuales participan en la regulación de la infección viral.
En su camino evolutivo de adaptación al hospedero humano, el SARS-CoV-2 genera diversos cambios genómicos. Entre ellos encontramos las mutaciones puntuales, que cambian una sola de las bases del genoma, y las inserciones y deleciones (llamadas también “indels”), que agregan o quitan bases. Estos cambios ocurren en todo el genoma, incluso en los genes accesorios u ORFs, los cuales participan en la regulación de la infección viral.
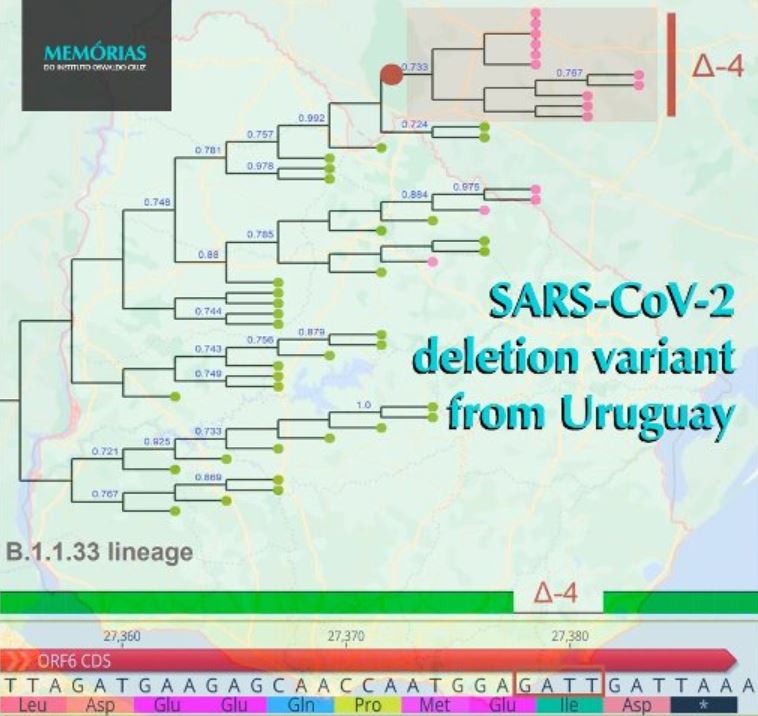
En este trabajo se reporta la emergencia y diseminación de una variante que presenta una deleción de 4 nucleótidos en el ORF6, un gen interesante porque codifica una proteína con actividad inmunomodulatoria. A pesar de ser una deleción, este cambio incrementa la información genómica, ya que cambia la terminación de la proteína, resultando en la adición de 2 aminoácidos.
Estos virus fueron detectados inicialmente en un brote ocurrido en un centro de salud de Montevideo en 2020 y posteriormente en otros casos de COVID-19 no relacionados epidemiológicamente. Se obtuvo la secuencia completa de sus genomas, utilizando secuenciación de nueva generación (NGS) en la Plataforma Genómica de Facultad de Ciencias.

Los datos epidemiológicos y de secuencia, así como la presencia de la deleción, indican que estas variantes de SARS-CoV-2 comparten un ancestro común, dando lugar a lo que se denomina un “clúster de transmisión”. El trabajo publicado muestra que los “indels” son marcadores fuertes y estables, y son sumamente útiles para el análisis y rastreo de los brotes epidémicos, particularmente cuando las secuencias genómicas son similares.
Además de la importancia epidemiológica, el análisis de la variabilidad genómica de SARS-CoV-2 nos enseña sobre los mecanismos básicos de variabilidad genética que el virus utiliza para evolucionar. La deleción observada se produce en una región repetida y podría haberse generado por una alteración de la replicación viral.
Este trabajo surge de la colaboración entre investigadores de las Secciones Genética Evolutiva y Virología de la Facultad de Ciencias con el Centro de Referencia de Influenza y otros virus Respiratorios (Depto. de Laboratorios de Salud Pública, MSP) y contó con la financiación de la Fundación Manuel Pérez.
Autores: Yanina Panzera#, Natalia Ramos#, Lucía Calleros, Ana Marandino, Gonzalo Tomas, Claudia Techera, Sofía Grecco, Sandra Frabasile, Eddie Fuques, Leticia Coppola, Natalia Goñi, Viviana Ramas, Cecilia Sorhouet, Victoria Bormida, Analia Burgueño, Maria Brasesco, Maria Rosa Garland, Sylvia Molinari, María Teresa Pérez, Rosina Somma, Silvana Somma, María Noelia Morel, Cristina Mogdasy, Héctor Chiparelli, Juan Arbiza, Adriana Delfraro*, Ruben Pérez*.
# igual contribución
*autores de correspondencia
DOI: 10.1590/0074-02760210275
Foto: Raul Echeverría para Facultad de Ciencias
